将耗时20分钟的复杂病理切片分析缩短至仅需1分钟,且无需医生进行繁琐的逐一人工标注。近日,一项由西安交通大学计算机科学与技术学院李辰教授团队联合剑桥大学完成的AI医学突破,让这一场景成为现实。该技术在国际上率先攻克了弱监督学习下数字病理全玻片(WSI)难以实现精确空间量化的瓶颈,被形象地比喻为病理诊断的“声纳系统”。这意味着,AI病理诊断正从单纯的“粗略分类”正式迈向“精准量化”的新时代。
在癌症精准诊疗中,千兆像素级的数字病理切片是“金标准”。然而,长期以来计算病理学面临着一个巨大的“黑盒”难题:现有的主流AI方法(多实例学习MIL)虽然能以较低成本判断切片“是否有癌症”,但无法回答“病灶具体在哪里”“恶性程度如何分布”以及“各类亚型占多大比例”。要想获得这些关键信息,往往需要病理医生投入大量精力进行逐像素的“手动描绘”,这在临床上几乎是不可能完成的任务。
针对这一难题,研究团队提出了名为SMMILe的新型人工智能框架,相关论文近日发表于国际肿瘤学权威期刊《自然·癌症》 (Nature Cancer) 。SMMILe是首个能在仅使用简化“病人级诊断标签”的情况下,实现对全玻片病灶进行精确空间量化的AI系统。SMMILe打破了传统弱监督算法“重分类、轻定位”的局限,在无需昂贵人工标注的条件下,能够像绘制地图一样,自动推断出肿瘤在组织中的具体位置、边界范围及不同亚型的空间分布。

为何SMMILe能做到其他AI做不到的事? 该技术的创新之处在于其独特的设计理念——它类似于一套用于图像的“声纳”系统,本质上赋予了计算机“在黑暗中视物”的能力。
传统方法往往因为缺乏详细坐标信息而“抓瞎”,或者只能捕捉到最明显的特征。而SMMILe通过融合特征压缩、参数自适应处理及马尔可夫随机场约束等前沿数学模型,能够敏锐捕捉到微弱的病理信号。即便是在没有任何位置标注信息的情况下,它也能像声纳探测海底地形一样,精准锁定并还原出具有生物学意义的肿瘤空间图谱。这不仅解决了AI模型的可解释性问题,更让病理分析的效率实现了数量级的飞跃——一张复杂的组织切片,人工分析可能耗时20分钟,而SMMILe仅需约1分钟即可生成详尽的量化报告。
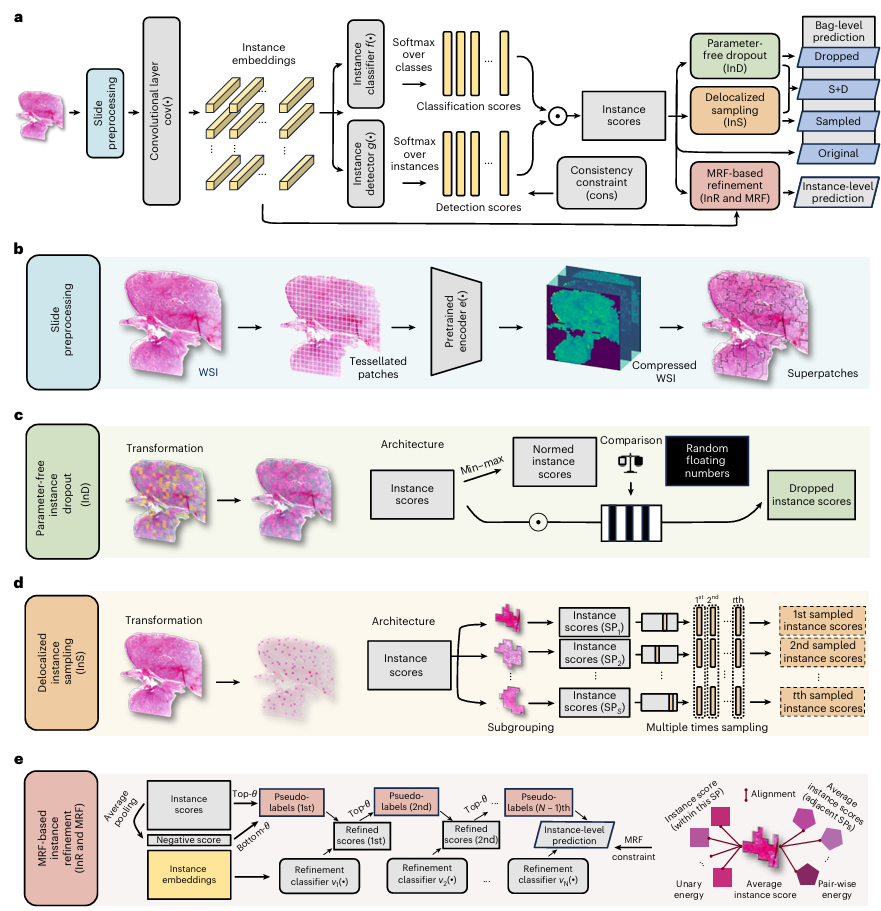
图1 SMMILe的模型结构
在具体评估中,研究团队基于涵盖乳腺、肺、卵巢、肾、胃和前列腺等六种癌症的3850张全切片,对SMMILe与九种当前主流计算病理学方法进行了系统比较。结果表明,在切片级分类任务中,SMMILe在各数据集上的整体表现达到或优于现有方法;在更具挑战性的病灶空间量化任务中,其在多个数据集上的空间AUC接近或超过90%,空间F1相较最佳对比方法最高可提升20个百分点以上。即便在采用病理基础模型等更强特征编码器、整体性能普遍抬升的条件下,SMMILe仍在多项空间指标上保持明显优势,尤其在多标签等复杂任务中表现突出。
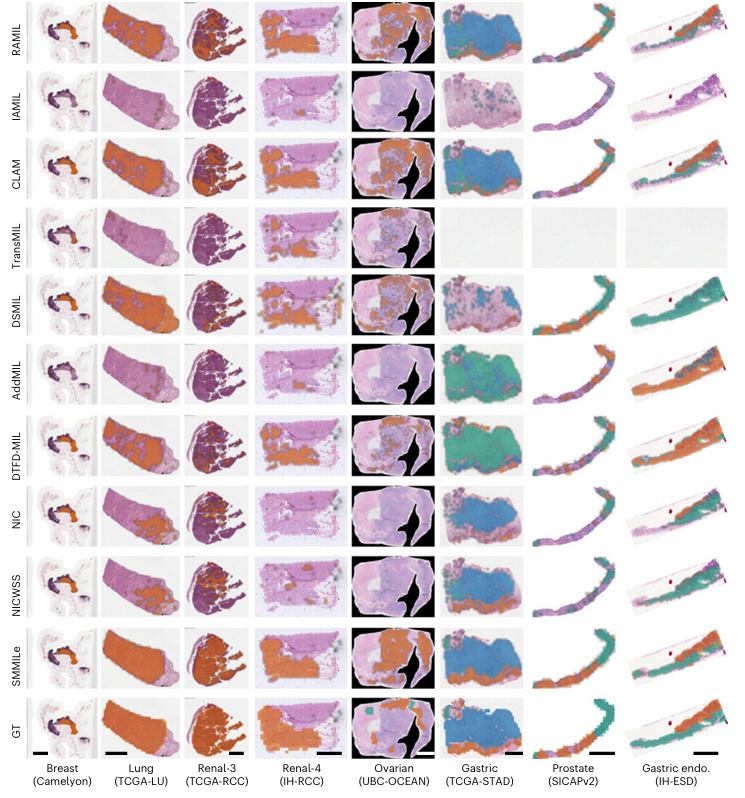
图2 SMMILe与其他方法的空间量化结果可视化对比
SMMILe的潜力不仅体现在诊断性能的提升。通过生成可靠的空间量化图谱,该方法为病理医生提供了更直观的组织结构信息,有助于在复杂病例中更快速、准确地锁定关键区域,确保患者能尽早获得最佳治疗方案,同时也为科研人员系统研究肿瘤异质性、探索不同组织亚型与预后、免疫反应及药物敏感性之间的关系提供了有力工具。未来,这一框架还有望进一步扩展,用于推断肿瘤的分子特征,将组织形态学与多组学数据紧密结合,推动综合癌症医学和精准医疗的发展。
该研究由西安交通大学计算机科学与技术学院李辰教授团队与剑桥大学等机构合作完成。论文第一作者为高泽宇博士,他于西安交通大学李辰教授团队获得博士学位后,在剑桥大学肿瘤学系继续从事医学人工智能研究工作。联合作者中,来自西安交通大学一附院病理科的主治医师王春宝参与了病理数据的整理与专业病理学支持。通讯作者包括西安交通大学李辰教授、龚铁梁副教授,以及剑桥大学肿瘤学系助理教授、早期癌症研究团队负责人Mireia Crispin-Ortuzar博士。研究工作获得英国癌症研究中心(Cancer Research UK)、马克癌症研究基金会(The Mark Foundation for Cancer Research)、英国国家健康研究院(NIHR)等机构的资助支持。
论文链接:https://www.nature.com/articles/s43018-025-01060-8
李辰教授团队主页:https://chenli.group/