肿瘤组织具有高度异质性,细胞类型多样、基因表达杂乱无章,区域界限模糊,传统的空间转录组技术难以全面揭示肿瘤的特性。传统方法通常将采样点视为独立的“房子”,统计每个点的细胞类型和基因表达信息,容易产生误差。为了解决这一难点问题,西安交通大学叶凯教授团队提出以基因表达的空间分布来构建整体计算框架,相关研究成果以“STMiner:以基因为中心的空间转录组学解析肿瘤组织”(STMiner: gene-centric spatial transcriptomics for deciphering tumor tissues)为题,于2025年2月12日发表在《细胞·基因组学》(Cell Genomics)杂志上。
叶凯教授团队长期致力于生物信息学方法研究,并将开发的方法运用于遗传病和肿瘤的早诊和精准治疗。由于肿瘤组织的高异质性和空间转录组数据的高稀疏性,传统侧重局部临近特征的策略往往在肿瘤空间转录组数据分析中出现遗漏和偏差。因此,叶凯教授团队开发了STMiner方法,利用二维高斯混合模型和最优传输理论,将离散的采样点转化为连续的基因分布地图,就像从零散的“房屋信息”中绘制出一张完整的“居民分布图”。这种方法不仅能更准确地反映肿瘤组织的真实情况,还能减少干扰信息,帮助科学家更清晰地观察基因的空间分布规律。凭借这一技术,研究团队成功定位了高度复杂边界不清的肿瘤组织中的独特基因表达模式,清晰区分了正常与异常组织的边界,并识别出一些此前被忽视的关键基因和空间结构。这些发现为肿瘤研究开辟了全新路径,有望为未来的精准治疗提供重要依据。
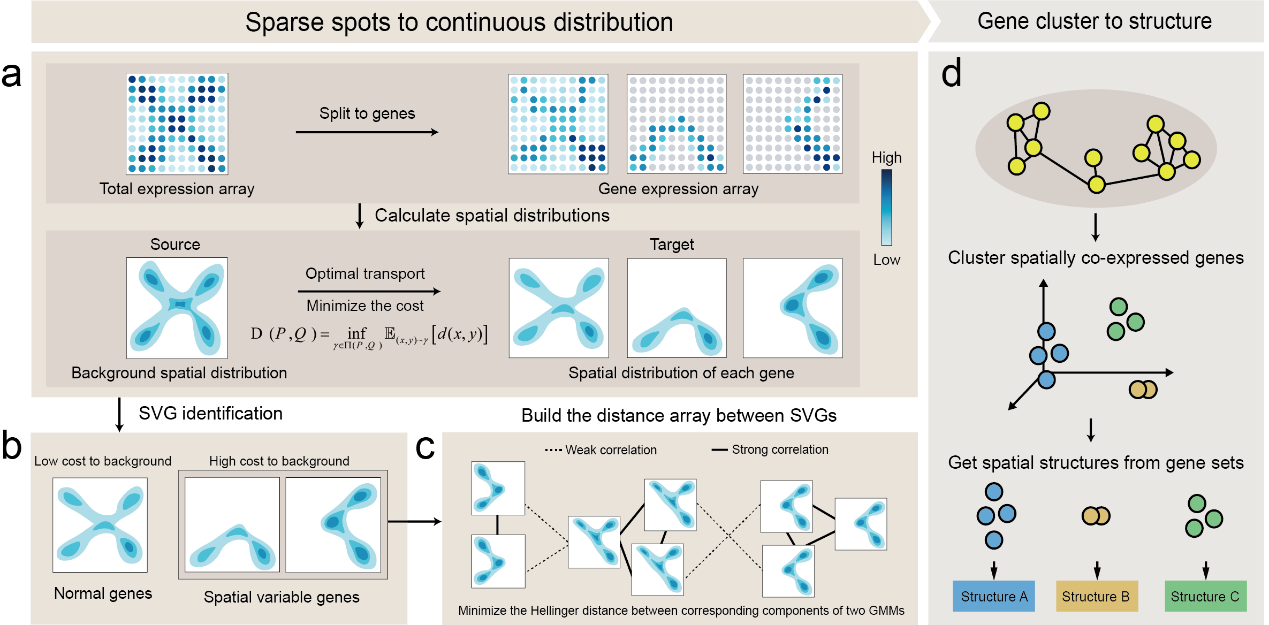
STMiner计算策略示意图
此项工作是西安交通大学信息与生物医学研究中心多学科交叉协作的又一重大成果,中心立足“四个面向”开展“理-医-工”交叉研究,组建了自动化、计算机、数学、生物、医学等多学科优势相融合的科研团队,打造“测-算-控”全链条研究体系,近年来在《科学》《自然》《自然·方法》《自然·生物技术》《自然·微生物学》等国际顶级期刊发表了一系列重要研究成果。得益于该协作模式,电信学部自动化科学与工程学院Stephen J. Bush教授在组学数据分析方面给予了重要指导;第一附属医院贾鹏博士、王澄瑶博士在算法验证、器官芯片构建方面提供了支持;电信学部计算机学院杨晓飞教授在算法开发方面提供了建议。
本论文的第一作者为电信学部自动化学院博士生孙培森,叶凯教授为通讯作者。研究得到了科技部重点研发专项、国家自然科学基金杰出青年基金等项目的支持。
论文链接:https://www.cell.com/cell-genomics/fulltext/S2666-979X(25)00027-8
西安交通大学信息与生物医学交叉研究中心团队主页:https://gr.xjtu.edu.cn/en/web/kaiye